Zespół Biotechnologii
Skład osobowy
- dr hab. Katarzyna Mikołajczyk
- dr Marcin Matuszczak
- dr Agnieszka Łopatyńska
- dr Joanna Wolko
- mgr Joanna Nowakowska
- Katarzyna Kozłowska
- mgr inż. Julia Żok
Tematyka badań
- Mapowanie chromosomów rzepaku ozimego za pomocą markerów DNA i poszukiwanie ich sprzężenia z cechami jakościowymi.
- Opracowanie i zastosowanie markerów genetycznych i funkcjonalnych do selekcji i hodowli przy użyciu markerów (MAS oraz MAB) nowych linii hodowlanych rzepaku ozimego.
- Marker genetyczny typu SCAR dla genu restorera Rfo w systemie męskiej sterylności typu ogura
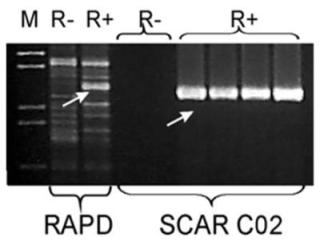 Marker typu SCAR
Marker typu SCAR
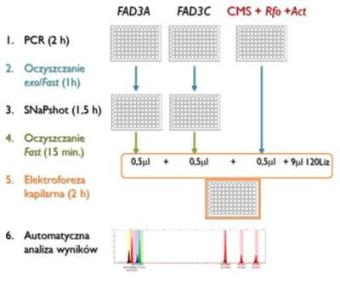
- Test genetyczny typu multipleks PCR, do identyfikacji linii oraz rekombinantów posiadających gen restorer Rfo a także męsko-sterylną cytoplazmę CMS w hodowli mieszańców F1 z wykorzystaniem systemu ogura
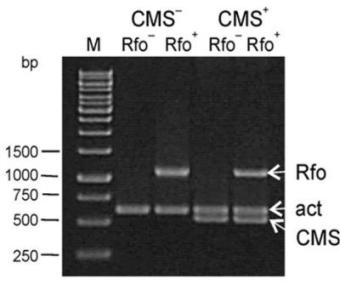 Test genetyczny typu multipleks PCR
Test genetyczny typu multipleks PCR
- Allelo-specyficzne markery funkcjonalne typu SNaPshot, do identyfikacji niezmutowanych i zmutowanych alleli genów desaturaz FAD3 w genomach A i C rzepaku, w liniach niskolinolenowych mutantów oraz ich rekombinantów z liniami podwójnie ulepszonymi
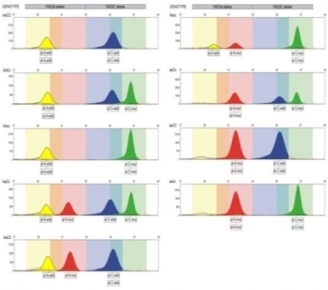 Allelo-specyficzne markery funkcjonalne typu SNaPshot
Allelo-specyficzne markery funkcjonalne typu SNaPshot
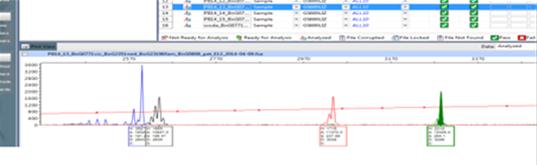
- Test genetyczny typu fluorescencyjny multipleks PCR, do monitorowania genu restorera Rfo, męsko-sterylnej cytoplazmy typu ogura oraz form allelicznych genów FAD3 w genomach A i C rzepaku w programach hodowli twórczej nowych linii rzepaku charakteryzujących się wysoką plennością oraz zmienionymi proporcjami kwasów tłuszczowych w oleju nasion
 Test genetyczny typu fluorescencyjny multipleks PCR
Test genetyczny typu fluorescencyjny multipleks PCR
- Identyfikacja markerów mikrosatelitarnych charakterystycznych dla różnych form hodowlanych rzepaku (fingerprinting) w celu ich zastosowania do określania zróżnicowania genetycznego form rodzicielskich dla poszerzania zmienności genetycznej oraz, w planowanych dalszych badaniach, monitorowania rearanżacji genomów podczas krzyżowania
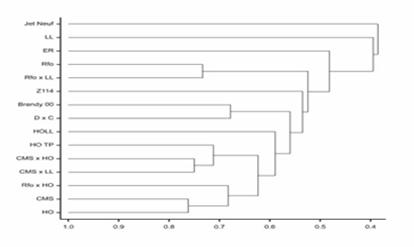 Identyfikacja markerów mikrosatelitarnych
Identyfikacja markerów mikrosatelitarnych
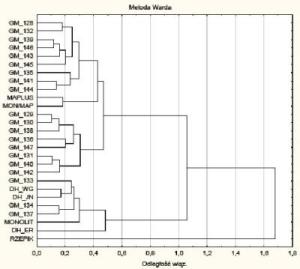
- Dystans genetyczny oceniany przy pomocy markerów molekularnych i jego wykorzystanie w hodowli odmian mieszańcowych.
- Zastosowanie markerów molekularnych do opracowanie metodyki hodowli odmian mieszańcowych.
- Badania asocjacyjne (genome wide association studies, GWAS) z wykorzystaniem najnowszych narzędzi bioinformatycznych: sekwencjonowanie nowej generacji (Illumina), genotypowanie metodami sekwencjonowania w celu identyfikacji genów odpowiedzialnych za cechy jakościowe rzepaku.
- Wykorzystywanie podwojonych haploidów (DH) uzyskiwanych metodą kultury izolowanych mikrospor w badaniach genetycznych i hodowli rzepaku ozimego:
- mapowanie chromosomów rzepaku ozimego za pomocą markerów molekularnych powiązanych z cechami jakościowymi nasion;
- tworzenie populacji linii homozygotycznych z genem restorerem dla hodowli odmian mieszańcowych rzepaku ozimego w systemie CMS ogura;
- ocena fenotypowa, określanie zdolności plonotwórczych linii DH rzepaku ozimego;
- badania nad zawartością w nasionach rzepaku tłuszczu, kwasów tłuszczowych, glukozynolanów, związków bioaktywnych (tokochromanole, sterole, związki fenolowe);
- badania statystyczno-genetyczne niektórych cech ilościowych oraz interakcji
genotyp × środowisko.
 Produkcja podwojonych haploidów rzepaku Brassica napus L.
Produkcja podwojonych haploidów rzepaku Brassica napus L.
- Badania nad wykorzystaniem metod biotechnologicznych dla zwiększenia bioróżnorodności rzepaku ozimego (Brassica napus L.):
- wprowadzanie obcego DNA do komórek haploidalnych metodą bezpośredniej kokultury zarodków mikrosporowych z Agrobacterium tumefaciens;
- uzyskiwanie linii podwojonych haploidów rzepaku ozimego z transformowanych zarodków mikrosporowych;
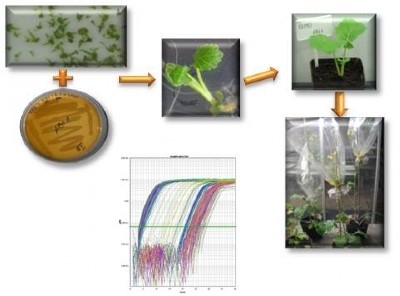 Transformacja genetyczna zarodków mikrosporowych
Transformacja genetyczna zarodków mikrosporowych
- resynteza Brassica napus poprzez krzyżowanie międzygatunkowe wybranych podgatunków Brassica rapa i Brassica oleracea;
- analiza embriologiczna, cytogenetyczna i molekularna resyntetyzowanych linii rzepaku ozimego;
- tworzenie linii DH z semi-RS rzepaku ozimego podwójnie ulepszonego otrzymanych poprzez krzyżowanie linii RS z rzepakiem podwójnie ulepszonym.
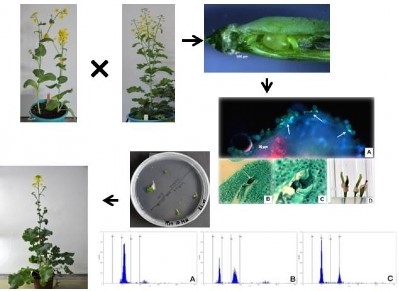 Resynteza rzepaku ozimego Brassica napus L.
Resynteza rzepaku ozimego Brassica napus L.
- Badania nad androgenezą in vitro gorczycy białej (Sinapis alba L) w kulturze izolowanych mikrospor.
 Androgeneza in vitro Sinapis alba L.
Androgeneza in vitro Sinapis alba L.